Servicios Personalizados
Revista
Articulo
Links relacionados
Compartir
Agrociencia (Uruguay)
versión impresa ISSN 1510-0839versión On-line ISSN 2301-1548
Agrociencia Uruguay vol.17 no.2 Montevideo dic. 2013
Caracterización de cepas de Xanthomonas citri sbsp. citri, agente causal del cancro cítrico
Russi Paola1, Menoni Mariana1, del Campo Raquel1, Peyrou Mercedes1
1Instituto de Investigaciones Biológicas Clemente Estable. Departamento de Biología Molecular. Avenida Italia 3318. 11600 Montevideo. Correo electrónico: mercepeyrou@gmail.com
Recibido: 28/8/12 Aceptado: 18/6/13
Resumen
Xanthomonas citri sbsp. citri (Xcc) es la especie bacteriana causante del cancro cítrico, enfermedad restrictiva para el comercio internacional de fruta fresca. Veinte aislamientos de Xcc, de Uruguay y la región, responsables de la forma más severa del cancro cítrico, fueron caracterizados a nivel fenotípico y genotípico con el fin de conocer su diversidad. Su identidad fue confirmada por anticuerpos específicos, PCR e inoculación. Se evaluó la virulencia en plantas de pomelo y la sensibilidad a antibióticos. A nivel genotípico, las cepas fueron caracterizadas según el perfil plasmídico y las secuencias repetidas ERIC. Todas las cepas resultaron sensibles a estreptomicina y ampicilina, a excepción de la cepa 89.5ex, resistente a ampicilina. En el análisis de virulencia, expresada por el número de pústulas inducidas/cm2 de tejido vegetal, se observaron diferencias entre las cepas aunque no es posible agrupar los aislamientos por similitud de virulencia. El aislamiento 138.2 se destaca por desarrollar mayor cantidad de pústulas en todos los ensayos realizados. Se identificaron tres perfiles plasmídicos, siendo mayoritario el perfil conformado por dos plásmidos, presentes en el 65% de las cepas. La «huella dactilar» obtenida por ERIC es igual en 18 de los aislamientos analizados, aunque dos de ellos presentaron una banda diferencial de 1992 pb. A pesar de las diferencias mencionadas, la población estudiada mostró un alto nivel de homogeneidad, característica ya descrita para Xcc. Es interesante observar que el aislamiento con mayor virulencia fue el único en el cual se detectaron tres plásmidos.
Palabras clave: CANCRO CÍTRICO, XANTHOMONAS CITRI, ERIC, PERFIL PLASMÍDICO, ANTIBIÓTICOS
Summary
Characterization of Xanthomonas citri sbsp. citri Strains, the Causal Agent of Citrus Canker
Xanthomonas citri sbsp. citri (Xcc) is the bacterium that causes citrus canker, a disease restrictive for the international fresh fruit trade. In order to know its diversity, twenty Xcc isolates from Uruguay and the region, responsible for the most severe form of citrus canker, were studied for phenotypic and genotypic characterization. Its identity was confirmed by specific antibodies, PCR and inoculation. The virulence in grapefruit plants and the sensitivity to antibiotics were evaluated. At a genotypic level, the strains were characterized according to the plasmid profile and the repeated ERIC sequences. All the strains were resistant to streptomycin and ampicillin, strain 89.5ex was an exception, being resistant to ampicillin. The analysis of virulence, expressed by the number of induced pustules/cm2 of vegetal tissue, showed differences among the strains. However, it is not possible to group the isolates by the degree of similarity of the virulence. The 138.2 isolate stands out for the development of a greater number of pustules in all the assays. Three plasmid profiles were detected; the profile formed by two plasmids represented the majority, being present in 65% of the strains. The «fingerprint» obtained by ERIC is the same in 18 of the analysed isolates, even though two of them showed a differential band of 1992 bp. In spite of the differences mentioned, a high level of homogeneity was found in the population studied, a characteristic already described for Xcc. Interestingly, the most virulent isolate was the only one in which three plasmids were found.
Keywords: CITRUS CANKER, XANTHOMONAS CITRI, ERIC, PLASMID PROFILE, ANTIBIOTIC
Introducción
La bacteria Xanthomonas citri sbsp. citri (Xcc) –antes X.axonopodis pv. citri– es responsable de la forma más severa del cancro cítrico, enfermedad que afecta a todas las especies de cítricos (Brunings y Gabriel, 2003) ocasionando defoliación de la planta, daño y caída prematura del fruto, secado de ramas y debilitamiento general del árbol (Graham et al., 2004). Sin embargo, su principal impacto económico se debe a la regulación impuesta sobre el comercio de productos cítricos (Canteros, 2006).
Esta situación determina la necesidad de contar con herramientas para la detección del agente causal y su identificación precisa, así como conocer la variabilidad del patógeno en una región determinada. Establecer la variabilidad y diversidad de un patógeno permite desarrollar técnicas de detección capaces de reconocer todas sus variantes posibles, realizar estudios epidemiológicos y establecer métodos efectivos de control. La discriminación de razas y patovares de Xcc ha sido de particular importancia en el establecimiento del riesgo en programas de erradicación de la enfermedad, en Florida-EEUU y otras zonas afectadas (Schoulties et al., 1987).
Para la caracterización bacteriana se utilizan varias técnicas basadas en rasgos fenotípicos (Louws et al., 1999; Khoodoo et al., 2005). Sin embargo, estas características pueden tener baja reproducibilidad y no necesariamente reflejan las relaciones genéticas de los aislamientos bacterianos. En contraste, los métodos basados en ADN son más estables y proporcionan un resultado más preciso de las relaciones genéticas. Varias de estas técnicas han sido aplicadas para caracterizar cepas de Xcc (Civerolo, 1985; de Souza Carvalho et al., 2005; Pruvost, et al., 1992; Hartung, 1992; Cubero y Graham, 2002, 2004; Shiotani, et al., 2000; Goncalves y Rosato, 2000, 2002; Mohammadi et al., 2001). Los resultados obtenidos con la secuenciación de regiones de ARNr evidencian el alto grado de conservación de estas regiones en el género Xanthomonas inhabilitando dicha metodología para la diferenciación a nivel de razas o patovares (Hauben et al., 1997).
Si bien no se utilizan antibióticos para el control de Xcc, el estudio de la resistencia a estos agentes permite ampliar la caracterización de la bacteria e identificar marcadores biológicos aplicables a la selección en ensayos de conjugación o transformación. Vernière et al. (1994), detectaron una variante de Xcc con altos niveles de resistencia a antibióticos relacionados con las penicilinas. Asimismo, se observó que los determinantes genéticos de la resistencia al antibiótico estreptomicina, se encuentran ligados a aquellos que determinan la resistencia a cobre (Cooksey, 1990; Sundin y Bender, 1993). Este aspecto es de relevancia ya que el control de la enfermedad en el campo se basa casi exclusivamente en productos a base de cobre. Para el caso de Xcc se ha reportado la existencia de cepas resistentes a cobre en Misiones y Corrientes en Argentina (Canteros, 1999), no así en Brasil (Maciel et al., 1998) hasta el momento.
En Uruguay, el cancro cítrico fue descrito en 1949, sobre plantas de limón, presentando una baja severidad y extensión. Estudios realizados por el Ministerio de Ganadería, Agricultura y Pesca, determinaron que se trataba de la cancrosis tipo B, producida por Xanthomonas citri sbsp. aurantifolli. En años posteriores, el ingreso de la bacteria causante del tipo A (Canale y Francis, 1988), ya presente en Brasil y Paraguay, determinó la rápida dispersión de la enfermedad debido a la mayor severidad de este tipo bacteriano. A partir de 1977 las autoridades fitosanitarias implementan diferentes medidas para su control que incluyen campañas de erradicación a nivel nacional (Arocena et al., 1979), inclusión de la enfermedad en esquemas de certificación de material de propagación, y determinación de zonas libres de la enfermedad que permiten cumplir con las exigencias de los mercados compradores. Distintos relevamientos y monitoreos realizados en años subsiguientes no han logrado aislar de muestras de campo la cepa B, encontrándose presente exclusivamente la cepa A.
Se ha observado una importante variación anual en la incidencia de la enfermedad, atribuible a factores dependientes de la planta como estado fisiológico y grado de madurez de los tejidos (Gottwald y Graham, 1992), el tiempo en que la hoja permanece húmeda y factores ambientales que favorecen dicha manifestación (Dalla Pria et al., 2006), aunque podría deberse a diferencias en la virulencia de las cepas predominantes. Sin embargo, no se han desarrollado estudios sobre la diversidad de la cepa A causante de la enfermedad en las distintas regiones citrícolas de Uruguay. El objetivo de este trabajo es obtener información sobre la diversidad genotípica y fenotípica de aislamientos de Xcc de nuestro país y de la región.
Materiales y métodos
Aislamientos Xcc
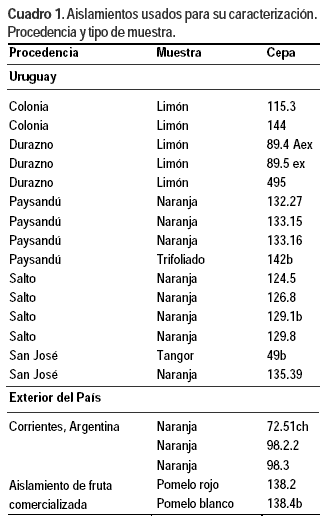
Los 20 aislamientos de Xcc se obtuvieron a partir de frutos, hojas y varetas de distintas especies cítricas, sintomáticas o asintomáticas, colectadas de diferentes regiones de Uruguay y de fruta comercializada en el mercado (Cuadro 1). El órgano vegetal asintomático o la lesión característica previamente aislada, se lavó en etanol 70% y se desinfectó con hipoclorito (3% cloro activo) durante 3 min. Se enjuagó nuevamente en etanol 70% y dos veces en agua destilada. El aislamiento de la bacteria se realizó por exudado de la población bacteriana presente en las muestras en tampón PBS 1X (8 g.L-1 NaCl, 0,2 g.L-1 KCl, 1,44 g.L-1 Na2HPO4, 0,24 g.L-1 KH2PO4) estéril, y posterior sembrado en placa con medio NB agar modificado (23 g.L-1 Nutrient broth, Oxoid, 1 g.L-1 glucosa, 15 g.L-1 agar) (del Campo et al., 2009) al cual se adicionaron kasugamicina (16 mg.L-1), cefalexina (16 mg.L-1) y clorotalonil (16 mg.L-1) como agentes selectivos (Timmer, 1988). Luego de su incubación a 28 ºC durante 48 h, los aislamientos que desarrollaron colonias típicas del género Xanthomonas –circulares, convexas, mucoides, brillantes y amarillas (Xanthomonas axonopodis pv citri, 2005)– fueron conservadas en medio NB con 30% de glicerol, a -80 ºC.
Identificación de los aislamientos por serología, amplificación de secuencias específicas y patogenicidad
Para la identificación serológica se realizaron ensayos de ELISA con anticuerpos monoclonales y policlonales específicos para Xcc. Se utilizó el kit de ELISA indirecto con anticuerpos monoclonales de AGDIA™, y el producido por la DGSA-MGAP para ELISA-DAS basado en anticuerpos policlonales obtenidos contra un aislamiento de Xcc de nuestro país, los dos anticuerpos son específicos para el tipo A de la bacteria, y la detección se evidencia por la acción de la fosfatasa alcalina conjugada sobre p-nitro fenil fosfato como sustrato. El valor límite de la absorbancia medida a 405 nm desarrollada por el sustrato, por encima del cual se considera un resultado positivo, se determinó por el doble del valor promedio de muestras no Xcc incluidas en el análisis.
La muestra se preparó a partir de un cultivo líquido de 107ufc.mL-1, precipitado por centrifugación a 3000 g y resuspendido en tampón carbonato (15 mM Na2CO3, 35 mM NaHCO3, pH 9.2), del que se dispensaron 100 µL de cada muestra en la microplaca. Como control positivo se utilizó el aislamiento 49b de Xcc caracterizado en el laboratorio (del Campo et al., 2009), como control negativo se incluyeron muestras de Xanthomonas fragariae y Xanthomonas campestris ambas procesadas de la misma forma que las muestras en estudio, y tampón carbonato como control de la reacción. Las muestras y los controles fueron analizados por duplicado.
Para la amplificación de secuencias específicas de Xcc se usaron los cebadores 2/3 descritos por Hartung et al. (1993), que amplifican una región presente en ambos plásmidos de la bacteria, y los cebadores pth1 y pth2 que hibridan con la señal de localización nuclear del gen de virulencia pthA, también presente en ambos plásmidos (Cubero y Graham, 2002) (Cuadro 2).
Para determinar la especificidad del ensayo de PCR con los cebadores 2/3 se incluyó una cepa de Pantoea agglomerans aislada de tejidos de cítricos.
Las condiciones de amplificación para los cebadores 2/3 incluyen desnaturalización inicial a 95 ºC durante 2 min y 30 ciclos de 30 s a 94 ºC, 1 min a 56 ºC y 45 s a 72 ºC, con una extensión final de 5 min a 72 ºC. Las condiciones de amplificación para los cebadores pth1/pth2 fueron 40 ciclos de 30 s a 94 ºC, 30 s a 58 ºC y 45 s a 72ºC, una desnaturalización inicial de 5 min a 94 ºC, y una extensión final de 10 min a 72 ºC. Las concentraciones de reactivos utilizadas para ambos pares de cebadores fueron 1X tampón Taq con (NH4)2SO4, 3 mM MgCl2, 0.1 µM de cada cebador, 200 µM de cada dNTPs y 1 U de polimerasa Taq (Fermentas™) en un volumen final de 50 µL. Para los ensayos de PCR se utilizó el termociclador Quatro TC-40. Los productos de PCR fueron separados por electroforesis en gel de agarosa al 2% en tampón TAE (Tris-acetato 40mM, EDTA 1 mM pH 8), teñido con bromuro de etidio (1 µg mL-1) y visualizados bajo luz UV.
La confirmación de la identidad por patogenicidad en citrus, de los diferentes aislamientos se realizó partiendo de una colonia de Xcc (aproximadamente 107 ufc) crecida en NB agar modificado y resuspendida en 1 mL de PBS 1X. Esta suspensión fue inoculada por duplicado en la cara abaxial de la hoja por infiltración con jeringa sin aguja (Siciliano et al., 2006), en hojas de pomelo inmaduras completamente expandidas (Verniére et al., 1998). Las plantas fueron mantenidas a 28 °C con 16 h de luz hasta la aparición de los síntomas característicos inducidos por Xcc.
Caracterización fenotípica por sensibilidad frente a antibióticos y virulencia
La sensibilidad de los 20 aislamientos de Xcc frente a ampicilina (10 µg) y estreptomicina (10 µg) fue evaluada utilizando la técnica del antibiograma o método de disco difusión descrita por Bauer et al. (1966). Alícuotas de cultivo de los distintos aislamientos con crecimiento entre 1x108 y 2x108 ufc mL-1, cuantificado por el método de turbidez de McFarland, se sembraron por triplicado en placas con medio agar Mueller Hinton (20g medio MH y 17g agar). Sobre la superficie de la placa se depositaron discos de papel de filtro impregnados con los antibióticos y se incubaron por 48 h a 28 ºC hasta visualizar crecimiento del tapiz bacteriano donde se midió el halo de inhibición de crecimiento.
Para evaluar la virulencia de los diferentes aislamientos de Xcc se inocularon por triplicado suspensiones con poblaciones bacterianas entre 103 y 104 ufc mL-1, de cada una de las cepas, por el método descrito. Con la aparición de las lesiones, estas fueron fotografiadas con una escala que permitió calcular el área infiltrada con el programa GIMP 2.2. Se contaron las pústulas desarrolladas y se calcularon la cantidad de pústulas por cm2 de tejido inoculado.
Caracterización genotípica por perfiles plasmídicos y amplificación de secuencias repetidas (PCR-ERIC)
Extracción de ADN plasmídico: Para la extracción de ADN plasmídico fue utilizado el método descrito por Sambrook et al. (1989) con algunas modificaciones. Se partió de 3 mL de cultivo crecido con agitación durante 24 h a 28 ºC (DO600nm= de 0,3-0,4, correspondiente a 2,4 x 108 ufc mL-1). El cultivo precipitado por centrifugación, lavado en agua destilada, se resuspendió en 100 µL de solución de lisozima (10 mg mL-1 de lisozima, 50 mM de glucosa, 25 mM de TrisHCl pH 8, 10 mM de EDTA pH 8), donde se incubó por 5 min a temperatura ambiente. Luego del agregado de 200 µL de solución de lisis (0,5% SDS, 0,2N de NaOH), se mezcló suavemente e incubó en hielo por 10 min. Se agregaron 150 µL de la solución 3 M de acetato de potasio pH 4,8 (Sambrook et al., 1989) y se centrifugó por 5 min a 12000 g. El sobrenadante fue transferido a un tubo nuevo al cual se le adicionó igual volumen de fenol: cloroformo:alcohol isoamílico (25:24:1). Centrifugado durante 5 min, la fase superior fue transferida a otro tubo y el ADN plasmídico precipitado con etanol absoluto y lavado con etanol 70%. El pellet de ADN se resuspendió en 50 µL de tampón TE (10 mM de Tris-HCl pH 8 y 1 mM de EDTA pH8) y se agregó ARNasa (10 µL mL-1).
La determinación de los perfiles plasmídicos se llevó a cabo por medio de una electroforesis en gel de agarosa 0,7% en buffer TAE 1X (40 mM Tris-acetato, 1 mM de EDTA, pH 8), durante 6 h a voltajes entre 70v y 90v. Para visualizar las bandas el gel fue teñido con bromuro de etidio (1 µg mL-1) durante 10 min y observado bajo luz UV. Se utilizaron como marcadores moleculares plásmidos de la cepa E.coli 39R861, de 166,6, 71,4, 40,6 y 7,8 kb (Jackson et al., 1999). Los tamaños moleculares de los plásmidos fueron estimados utilizando el software Kodak 1D Image Analysis a partir de la movilidad relativa de cada banda experimental comparada con las bandas de referencia de la cepa E.coli 39R861.
La diversidad genética de las cepas de Xcc fue analizada por medio de la amplificación de secuencias repetidas ERIC (enterobacterial repetitive intergenic consensus) (Hulton et al., 1991). Para ello se utilizaron los cebadores ERIC1 y ERIC2 (Cuadro 2) según las condiciones descritas por Cubero y Graham (2002). La extracción de ADN genómico se realizó con el Kit comercial Sigma™ (GeneElute bacterial genomic DNA kit). La concentración de reactivos utilizada fue 1X de tampón Taq, 3 mM de MgCl2, 0,6 µM de cada cebador, 200 µM de cada dNTPs y 2 U de Taq polimerasa (Fermentas™), en un volumen final de 25 µL. El programa de amplificación fue de 40 ciclos de 30 s a 94 ºC, 30s a 52 ºC y 1 min a 72 ºC, con una desnaturalización inicial de 50 min a 94 ºC y un paso de extensión final de 10 min a 72 ºC. Los productos de PCR fueron separados por electroforesis en gel de agarosa 1,5% en tampón TAE. Luego de teñido con bromuro de etidio (1 mg.mL-1) durante 10 minutos se visualizaron los fragmentos de ADN con luz UV. El tamaño de las bandas diferenciales fue calculado con el programa Kodak 1D Image Analysis software utilizando como referencia el marcador GeneRuler 100pb de Fermentas™.
Resultados y discusión
Identificación de los aislamientos
La identificación serológica se realizó con dos anticuerpos específicos contra Xcc. Todas las cepas de Xcc desarrollaron coloración amarilla por la acción de la enzima conjugada sobre su sustrato, con valores de absorbancia a 405 nm superior al umbral calculado para los dos tipos de anticuerpos ensayados, mientras que las muestras correspondientes a los otros géneros bacterianos no desarrollaron color.
La amplificación de secuencias específicas del genoma de Xcc permitió la identificación molecular de los aislamientos. En todos los aislamientos analizados se obtuvo una banda de 222 pb con los cebadores 2/3 (Figura 1) y una banda de 197 pb con pth1/pth2 (Figura 2a y 2b). La cepa de Pantoea agglomerans no amplificó con los cebadores 2/3 (Figura 1), confirmando la especificidad de dichos oligonucleótidos.
Todos los aislamientos de Xcc confirmaron su identidad por la inducción de síntomas característicos de esta especie en hojas de pomelo, aproximadamente al séptimo día post-inoculación.
Caracterización de los aislamientos
Para analizar la virulencia, medida como pústulas/cm2 de tejido vegetal inoculado, se inocularon plantines de pomelo por triplicado, con una población entre 103 y 104 ufc mL-1 de todas las cepas separadamente y a diferentes diluciones en hoja de pomelo susceptibles. A pesar de observarse diferencias en la cantidad de pústulas desarrolladas por unidad de superficie por la distintas cepas y que existe una clara diferencia entre los aislamientos más agresivos –que indujeron mayor cantidad de pústulas por cm2 de tejido inoculado– y aquellos más leves, los resultados obtenidos no permitieron separar grupos claramente definidos de cepas más virulentas y menos virulentas. En función de estos primeros resultados se realizó un segundo ensayo en el cual se escogieron las cepas 144, 126.8, 115.3, 98.3, 98.2.2, 138.2, 132.27, 89.4Aex y 89.5ex que mostraron comportamientos diferentes en cuanto a la virulencia, en el ensayo preliminar.
Para minimizar la influencia que pudiera manifestar las variables dependientes de la planta e incluso el estado de desarrollo de la hoja en la expresión de síntomas, se utilizó la cepa 49b de Xcc como cepa de referencia que fue inoculada en la misma hoja que la cepa a evaluar. Como indicador de virulencia se utilizó la relación entre pústulas/cm2 desarrolladas por la cepa en estudio y por las desarrolladas por la cepa 49b. En trabajos previos se determinó que existió una relación lineal entre la población inoculada y el número de pústulas formadas cuando se trabajó con poblaciones bacterianas entre 103 y 104 ufc mL-1 (Stall et al., 1982). Para el análisis de varianza se utilizó el programa SAS 9.1, aplicando el procedimiento GLM, y las medias fueron separadas por la prueba «t « con una P=0,05. Del análisis estadístico de la cantidad de pústulas producidas por cada cepa en estudio, en tres repeticiones surge que, tanto en el ensayo preliminar como en el segundo ensayo, la cepa 138.2 produjo más pústulas/cm2 mientras que las cepas 126.8 y 89.5ex manifestaron la menor virulencia (Figura 3). El resto de las cepas analizadas mostró una virulencia intermedia de las cepas mencionadas, sin diferencia significativa entre ellas. Por lo tanto, con el método de inoculación utilizado, es posible apreciar diferencias en la capacidad de desarrollar síntomas, sin que podamos afirmar que ocurra lo mismo en infecciones naturales.
Los resultados de los antibiogramas muestran que las cepas de Xcc estudiadas fueron sensibles a ambos antibióticos: ampicilina y estreptomicina. La sensibilidad de las cepas fue similar para cada antibiótico y el único aislamiento que presentó resistencia a ampicilina fue la cepa 89. 5 ex (Figura 4). La ampicilina es un antibiótico del grupo de las penicilinas y la resistencia de Xanthomonas citri sbsp. citri a este tipo de antibiótico fue reportada por Vernière et al. (1994), en aislamientos obtenidos en las islas del Océano Índico. Con respecto a la estreptomicina, todas las cepas analizadas fueron sensibles. A pesar de que no se ha reportado resistencia a cobre en Uruguay, debido a la importancia que tiene el cobre en el control de la enfermedad en las plantaciones comerciales, la posibilidad de la aparición de resistencia debería ser monitoreada de forma de detectar la aparición de cepas que no puedan ser controladas con los tratamientos químicos usados de rutina. Si bien las cepas de este trabajo son todas sensibles a estreptomicina, no podemos afirmar que también sean sensibles a cobre, ya que no se evaluó dicha condición.

Caracterización genotípica
La caracterización plasmídica de cepas de Xcc estudiadas reveló la presencia de tres perfiles distintos (Cuadro 3), en donde 13 cepas tienen dos plásmidos cuyos tamaños son de 36 ± 5 kb y 81 ± 8 kb, seis tienen sólo un plásmido de 131 ± 9 kb y una cepa tiene tres plásmidos, de 36, 81 y 131 kpb. El tamaño de los plásmidos se estimó en base al promedio de los valores calculados por el software Kodak 1D Image Analysis a partir de tres corridas electroforéticas para cada muestra. En la Figura 5 a y b se muestran los perfiles plasmídicos de las 20 cepas, junto con los plásmidos de la cepa de E.coli 39R861 utilizados como referencia. Un 65% de las cepas presentan solo los plásmidos de 36 y 81 kb. Otros plásmidos de diferentes tamaños, no pudieron ser detectados con la metodología utilizada para la extracción de ADN. Sin embargo, en el trabajo de de Souza Carvalho et al. (2005) donde se analizaron varias cepas de Xcc de Brasil y algunas cepas de Bolivia, Paraguay, Argentina y Uruguay, se detectaron plásmidos de 57.7 kb, 60.8 kb, 68.1 kb, 72.6 kb y 83 kb. De las cepas que estudiaron, el plásmido de 83 kb estuvo presente en una cepa de Rio Grande do Sul, una cepa de Santa Catarina, una de Paraná, en dos cepas de Paraguay y en la única cepa de Uruguay de dicho trabajo. Es importante notar que estas regiones citrícolas son las geográficamente más cercanas a Uruguay. El hecho de que aislamientos provenientes de estas zonas tengan el plásmido de 83 kb, que podría ser equivalente al plásmido de 81± 8 kb detectado en el presente trabajo, sugiere que nuestras cepas podrían tener un origen común con las cepas del trabajo previamente nombrado.
El patrón de bandas obtenido con ERIC-PCR fue similar en casi todos los aislamientos (Figura 6 a y b). Sólo los aislamientos 98.3 y 89.4Aex presentaron una banda diferencial, cuyo tamaño molecular es de 1992 pb, aproximadamente (Figura 7). Se puede observar que el patrón de bandas obtenido para X. campestris y X. fragariae es claramente diferente al de Xcc (Figura 8). Si tenemos en cuenta los trabajos de Louws y colaboradores que sostienen que esta técnica es útil para una rápida caracterización molecular de bacterias patógenas de plantas, pudiendo diferenciar patovares, incluso cepas dentro de una misma especie (Louws, et al., 1999, 1994), podríamos afirmar que el análisis de la PCR-ERIC aplicado a distintos asilamientos de Xcc reveló que la mayoría de las cepas son homogéneas en este tipo de secuencia repetida, ya que se observó únicamente una banda diferencial en dos de ellas.
Con los resultados obtenidos podemos decir que el análisis de los ERIC no proporciona datos suficientes para la discriminación entre cepas de Xcc, aunque sí se encontraron diferencias en el patrón de bandas entre Xanthomonas fragariae, Xanthomonas campestris y Xcc, mostrando su capacidad de discriminar entre especies (Louws et al., 1999).
Conclusiones
Los resultados de este trabajo concuerdan con los alcanzados por otros investigadores y muestran poca variación entre los distintos aislamientos o cepas de Xcc, tanto en las características genotípicas como fenotípicas estudiadas.
Sin embargo, con las características analizadas, fue posible encontrar –aunque con escasa representación– algunos aspectos diferenciales en la población de Xcc estudiada, como la aparición de resistencia a antibióticos, diferencias en virulencia de las distintas cepas, y diferentes perfiles electroforéticos de secuencias repetidas.
Este trabajo permite concluir, que todas las cepas analizadas son capaces de ser detectadas e identificadas por las técnicas utilizadas tradicionalmente como técnicas serológicas y amplificación por PCR de secuencias específicas.
Teniendo en cuenta la correlación citada por otros autores entre la presencia de determinantes genéticos relacionados a la resistencia a estreptomicina y a cobre, sería necesario analizar la sensibilidad frente a diferentes diluciones de cobre con el fin evaluar si existe resistencia a este compuesto en la población bacteriana. En la evaluación de la sensibilidad a ampicilina, solo la cepa 89.5ex mostró resistencia por el método estudiado.
Del análisis de virulencia surgió que la cepa 138.2 es la más virulenta. Con el fin de confirmar los resultados, es necesario evaluar la virulencia por otros métodos, como por ejemplo teniendo en cuenta el diámetro y características de la pústula inducida (Shiotani et al., 2007).
De los tres perfiles plasmídicos encontrados, el que presenta dos plásmidos es el más representado, estando presente en trece de los aislamientos de Xcc. De las siete cepas restantes solamente una posee tres plásmidos, la 138.2. Los perfiles obtenidos por la amplificación de las secuencias repetidas ERIC revelaron una gran homogeneidad entre los aislamientos de Xcc. Sin embargo, dos cepas de distinta procedencia se distinguen del resto por la presencia de una banda diferencial. Los perfiles obtenidos con las otras dos especies de Xanthomonas se diferencian claramente de cualquiera de las obtenidas con los diferentes aislamientos de Xcc, lo que podría contribuir a su diferenciación.
En este trabajo no fue posible relacionar espacial o geográficamente los aislamientos mediante el análisis de las características evaluadas. Cabe destacar que la única cepa que mostró la presencia de tres plásmidos resultó ser la de mayor virulencia. Este resultado promisorio requerirá de mayor número de ensayos confirmatorios para establecer dicha asociación. También se debería realizar un estudio de los componentes genéticos contenidos en estos plásmidos y su posible contribución a la mayor agresividad observada.
El análisis futuro de secuencias BOX, REP, análisis de restricción, sensibilidad frente a otros antibióticos, resistencia a cobre, o el estudio de otras características, permitirá ampliar nuestro conocimiento sobre este patógeno, y estudiar la correlación de dichas características con su comportamiento en campo, contribuyendo a explicar algunas de las dificultades encontradas en el control del cancro cítrico.
Bibliografía
Bauer AW, Kirby MM, Sherris JC, Turck M. 1966. Antibiotic susceptibility testing by a standarized single disk method. American Journal of Clinical Pathology, 45: 493 – 496.
Brunings AM, Gabriel DW. 2003. Xanthomonas citri: breaking the surface. Molecular Plant Pathology, 4: 141-157.
Canale F, Francis M. 1988. Aplication of ELISA for the campaign to prevent and eradicate citrus canker in Uruguay. En: Proceedings of International symposium of citrus canker, declínio/blight and similar diseases. Campinas: Fundación Cargill. pp. 160-174.
Canteros BI. 2006. Management of citrus canker in Argentina : A review. En: Proceedings of the International Society of Citriculture: 10th International Citrus Congress ; 15 - 20 febrero 2004 ; Agadir, Morocco. s.l. : International Society of Citriculture. pp. 696-704.
Canteros BI. 1999. Copper resistance in Xanthomonas campestris pv. citri. En: Mahadevan A. [Ed.]. Plant pathogenic bacteria : Proceedings of the International Society of Bacteriology. Chennai : Centre for Advanced Study in Botany. University of Madras. pp. 455-459.
Civerolo EL. 1985. Indigenous plasmids in Xanthomonas campestris pv. citri. Phytopathology, 75: 524 -528.
Cooksey DA. 1990. Genetics of bactericide resistance in plant pathogenic bacteria. Annual Review of Phytopathology, 28: 201-219.
Cubero J, Graham JH. 2004. The leucine responsive regulatory protein (lrp) gene for characterization of the relationship among Xanthomonas species. International Journal of Systematic and Evolutionary Microbiology, 54: 429 -437.
Cubero J, Graham JH. 2002. Genetic relationship among worldwide strains of Xanthomonas causing canker in citrus species and design of new primers for their identification by PCR. Applied and Environmental Microbiology, 68: 1257-1264.
Dalla Pria M, Christiano RCS, Furtado EL, Amorin L, Bergamin Filho A. 2006. Effect of temperature and leaf wetness duration on infection of sweet oranges by Asiatic citrus canker. Plant Pathology, 55: 657-663.
de Souza Carvalho FM, Pereira L, Pereira R. 2005. Genetic diversity of Xanthomonas axonopodis pv. citri based on plasmid profile and pulsed field gel electrophoresis. Genetics and Molecular Biology, 28: 446 -451.
del Campo R, Russi P, Mara P, Mara H, Peyrou M, Ponce de León I, Gaggero C. 2009. Xanthomonas axonopodis pv. citri enters the VBNC state alter copper treatment and retains its virulence. FEMS Research Letters, 298: 143-148.
Goncalves ER, Rosato YB. 2002. Phylogenetic analysis of Xanthomonas species based upon 16S-23S rDNA intergenic spacer sequences. International Journal of Systematic and Evolutionary Microbiology, 52: 355-361.
Goncalves ER, Rosato YB. 2000. Genotypic characterization of xanthomonad strains isolated from passion fruti plants (Passiflora, spp.) and their relatedness to different Xanthomonas species. International Journal of Systematic and Evolutionary Microbiology, 50: 811-821.
Gottwald TR, Graham JH. 1992. A device for precise and nondisruptive stomatal inoculation of leaf tissue with bacterial pathogens. Phytopathology, 82: 930-935.
Graham JH, Gottwald TR, Cubero J, Achor DS. 2004. Xanthomonas axonopodis pv. citri: factors affecting successful eradication of citrus canker. Molecular Plant Pathology, 5: 1-15.
Hartung JS. 1992. Plasmid-based hybridization probes for detection and identification of Xanthomonas campestris pv. citri. Plant Disease, 76: 889-893.
Hartung JS, Daniel JF, Pruvost OP. 1993. Detection of Xanthomonas campestris pv. citri by the polymerase chain reaction method. Applied and Environmental Microbiology, 59: 1143-1148.
Hauben L, Vauterin L, Swings J, Moore ERB. 1997. Comparison of 16S ribosomal DNA sequences of all Xanthomonas species. International Journal of Systematic Bacteriology, 47: 328-335.
Hulton CSJ, Higgins CF, Sharp M. 1991. ERIC sequences: a novel family of repetitive elements in the genomes of Escherichia coli, Salmonella typhimurium and other enterobacteria. Molecular Microbiology, 5: 825-834.
Jackson RW, Athanassopoulos E, Siamis GT, Mansfield JW, Esma A, Arnold DW, Gibbon MJ, Murillo J, Taylor JD, Vivian A. 1999. Identification of a pathogenicity island, which contains genes for virulence and avirulence, on a large native plasmid in the bean pathogen Pseudomonas syringae pathovar phaseolicola. Proceeding of National Academie of Science, 96: 10875-10880.
Khoodoo MHR, Sahin F, Donmez MF, Jaufeerally-Fakim Y. 2005. Molecular characterisation of Xanthomonas strains isolated from aroids in Mauritius. Systematic and Applied Microbiology, 28: 366-380.
Louws FJ, Rademaker J LW, de Bruijn FJ. 1999. The three ds of PCR-based genomic analysis of phytobacteria: diversity, detection and disease diagnosis. Annual Review of Phytopathology, 37: 81-125.
Louws FJ, Fulbright DW, Stephens CT, Bruijn FJ. 1994. Specific genomic fingerprints of phytopathogenic Xanthomonas and Pseudomonas pathovars and strains generated with repetitive sequence and PCR. Applied and Environmental Microbiology, 60: 2286-2295.
Maciel JLN, Duarte V, Ayub MZ. 1998. Plasmid DNA restriction profile and copper sensitivity of Xanthomonas axonopodis pv. citri from Rio Grande do Sul, Brazil. Fitopatologia Brasileira, 23: 116-120.
MAP. 1979. Aspectos de la campaña de prevención y erradicación del cancro cítrico en Uruguay. Montevideo : Ministerio de Agricultura y Pesca. Dirección de Sanidad Vegetal. 25p. (Informe técnico ; 8).
Mohammadi M, Mirzaäee MR, Rahimian H. 2001. Physiological and biochemical characteristics of Iranian Strains of Xanthomonas axonopodis pv. citri, the causal agent of citrus bacterial canker disease. Journal of Phytopathology, 149: 65 -75.
Pruvost O, Hartung JS, Civerolo EL, Dubois C, Perrier X. 1992. Plasmid DNA fingerprint distinguish pathotypes of Xanthomonas campestris pv. citri, the causal agent of citrus bacterial canker disease. Phytopathology, 82: 485 -490.
Sambrook J, Fritsch EF, Maniatis T. 1989. Molecular cloning: a laboratory manual. New York : Cold Spring Harbor Press. 545p.
SchoultiesCL, Civerolo EL, Miller JW, Stall RE, Krass CJ, Poe SR, Ducharme EP. 1987. Citrus canker in Florida. Plant Disease, 71: 388-395.
Shiotani H, Fujikawa T, Ishihara H, Tsuyumu S, Ozaki K. 2007. A pthA homolog from Xanthomonas axonopodis pv. citri responsible for host-specific suppressión of virulence. Journal of Bacteriology 189: 3271-3279.
Shiotani H, Ozaki K, Tsuyumu S. 2000. Pathogenic interactions between Xanthomonas axonopodis pv. citri and cultivars of pummelo (Citrus grandis). Phytopathology, 90: 1383-1389.
Siciliano F, Torres P, Sendin L, Bermejo C, Filippone P, Vellice G, Ramallo J, Castagnaro A. 2006. Analysis of the molecular basis of Xanthomonas axonopodis pv. citri patogénesis in Citrus limon. Electronic Journal of Biotechnology, 9(3): 199 -204.
Stall RE, Marcó GM, Canteros BI. 1982. Importance of mesophyll in mature-leaf resistance to cancrosis of citrus. Phytopathology, 72: 1097-1100.
Sundin GW, Bender CL. 1993. Ecological and genetic analysis of copper and streptomycin resistance in Pseudomonas syringae pv. syringae. Applied and Environmental Microbiology, 59: 1018-1024.
Timmer LW. 1988. Evaluation of bactericides for control of citrus canker in Argentina. Proceedings of the Florida State Horticultural Society, 101: 6-9.
Verinère C, Hartung JS, Pruvost OP, Civerolo EL, Alvarez AM, Maestri P, Luisetti J. 1998. Characterization of phenotypically distinct strains of Xanthomonas axonopodis pv. citri from Southwest Asia. European Journal of Plant Pathology, 104: 477-487.
Vernière C, Pruvost O, Dubois C, Couteau A, Luisetti J. 1994. Variations among the strains of Xanthomonas isolated from citrus in the sensitivity to antibiotics. En: Lemattre SFM, Rudolph K, Swings JG. [Eds.]. Proceedings of the 8th International Conference on Plant Pathogenic Bacteria ; 9 - 12 junio 1992 ; Versailles, Francia. Versailles : INRA. (Colloques de l’INRA ; 66). pp. 247-251
Xanthomonas axonopodis pv. citri. 2005. EPPO Bulletin, 35(2): 289-294.